
이거 읽으면 인코에 바나나???!!!!! "노잼"은 도태되고 "예스잼"만 선발되어야 하나요?
현재 우리가 먹는 바나나는 대부분 캐번디시 품종으로, 1950년대 파나마병이 창궐했을 때 저항성이 없던 미셸 품종을 성공적으로 대체했으나 이후 발생한 신파나마병에는 취약하여 멸종 위기에 처했습니다. 호주 퀸즐랜드 공대 제임스 데일 교수 연구진은 야생 바나나(Musa acuminata)에서 신파나마병 저항성 유전자를 발견하고 유전자 편집을 통해 캐번디시 바나나에 해당 유전자를 추가하였습니다(Dale et al., 2017). 그 결과 기존 캐번디시의 우수한 품질에 더해 신파나마병 저항성까지 갖춘 슈퍼 바나나 품종이 탄생했고, 2024년 2월 호주에서 세계 최초 유전자 교정 품종에 대한 재배와 판매 허가까지 이루어 냈습니다. 재배원종 중 하나이기는 하지만 씨도 많고 시장 선호도 면에서는 가치가 없어 보였던, 즉 "노잼" 바나나였으나 데이터 분석을 통해 병 저항성 관련 유전자를 보유하고 있음이 밝혀져 유전자원으로서의 잠재력을 확인하게 해 준 사례입니다.
㈜인실리코젠에서는 현재 기준으로 "노잼"의 쓸모없는 자원도 미래 육종 목표에 따라 충분히 활용될 수 있는 무한한 가능성을 가지고 있음을 잘 알고 있습니다. 생물학적 데이터에 대한 이해를 바탕으로 데이터의 관리, 분석 및 활용에 이르기까지 데이터 기반 육종이 실현될 수 있도록 지속해서 노력하고 있습니다. 바나나의 사례와 같이, 다양한 작물의 유전자원으로서의 특성을 디지털화함으로써 빅데이터의 기반을 구축하고 생물정보 분석과 AI 기술 융합을 통하여 육종 분야의 새로운 가능성을 열어가고 있습니다. 데이터 육종의 동반자로서 저희의 이러한 도전과 결실을 소개해 드리고자 합니다.
ibreeder: 데이터 육종을 위한 All-in-One 플랫폼
ibreeder는 웹 기반 데이터 육종 플랫폼으로, 유전자원, 표현형 및 유전형 정보를 규격화하여 효율적으로 관리합니다(그림 1).
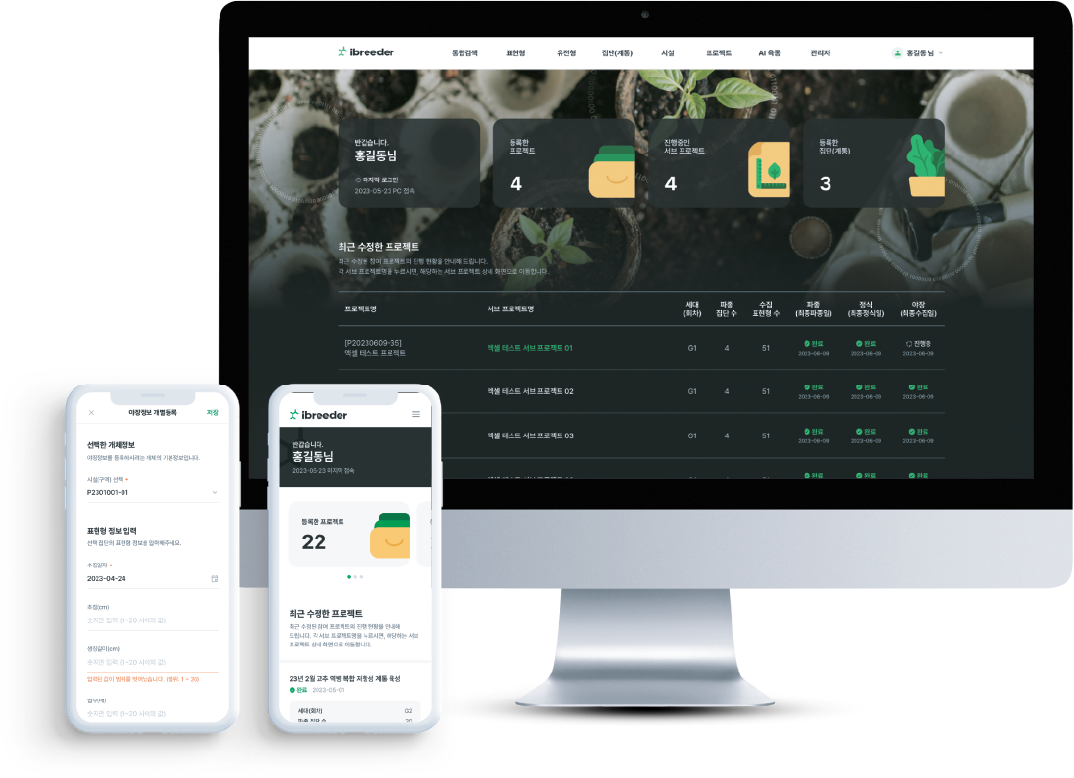
ibreeder의 주요 기능은 다음과 같습니다(그림 2).
디지털 야장: 작물의 유전자원, 원예 형질, 유전형 및 마커 정보를 간편하게 등록하고 관리할 수 있게 해 줍니다.
데이터 관리: 작물에 대한 모든 데이터를 정규화함으로써, 데이터의 수집 시점, 목적, 수집자가 다르더라도 모든 정보가 효과적으로 통합되어 사용자가 데이터를 쉽게 활용할 수 있습니다.
계통 관리: 통합 검색 기능을 통해 관심 원예 형질이 우수한 자원을 선발하여 교배조합을 작성할 수 있습니다. 이 과정에서 자손의 가계는 모부본의 가계에 따라 자동으로 업데이트되어 가계 관리가 훨씬 용이해집니다.
AI 육종: ibreeder만의 AI 기반 원예 형질 예측 기능을 통해 우수 자원을 선발하고, AI 교배조합 시뮬레이션을 사용하여 자손의 원예 형질을 예측할 수 있습니다. 이를 통해 교배 효율이 높은 모부본을 우선으로 선발할 수 있어, 육종 효율을 크게 향상할 수 있습니다.
데이터 보안: 모든 데이터는 사용자 권한 관리를 통해 보안이 유지되므로, 중요한 자원과 정보 유출에 대한 염려를 크게 줄일 수 있습니다.

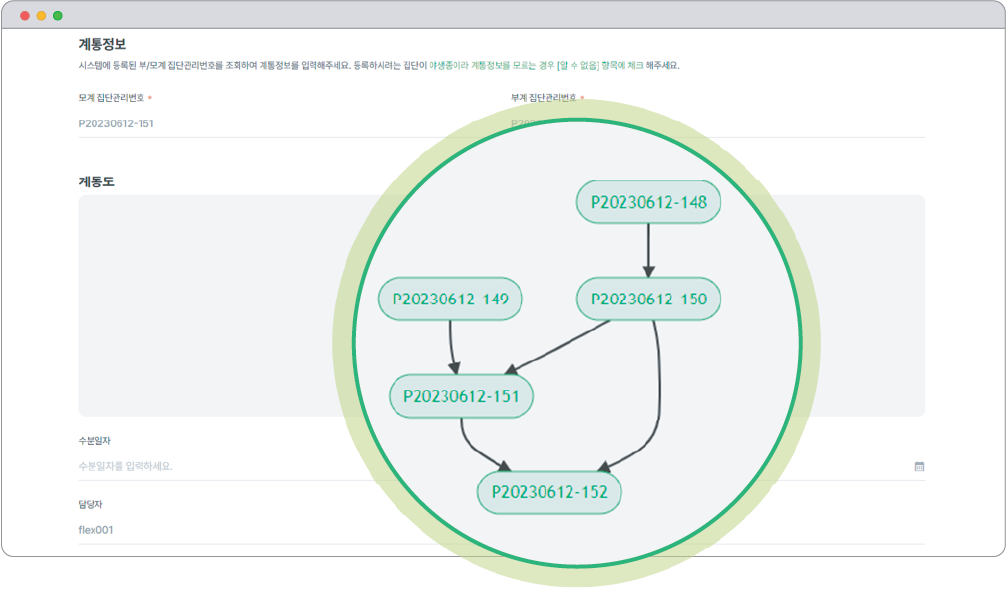
원예 형질이 뛰어난 우수 계통은 모부본으로서 특별히 관리됩니다. 육종 목표에 따라 계통 육성 이력(계통도)과 원예 형질 및 마커 정보를 근거로 모부본을 선발하고 교배 계획을 수립하는 데 도움을 줍니다(그림 3). 이때, AI 원예 형질 예측 및 교배조합 시뮬레이션 기능은 교배조합별 자손의 원예 형질을 예측함으로써 성공 확률이 높은 계통을 우선 육성할 수 있도록 돕습니다.
모든 교배조합별 파종, 가식, 정식 이력을 체계적으로 관리함으로써 자원의 효율적인 활용과 육종 과정의 투명성을 높일 수 있습니다.
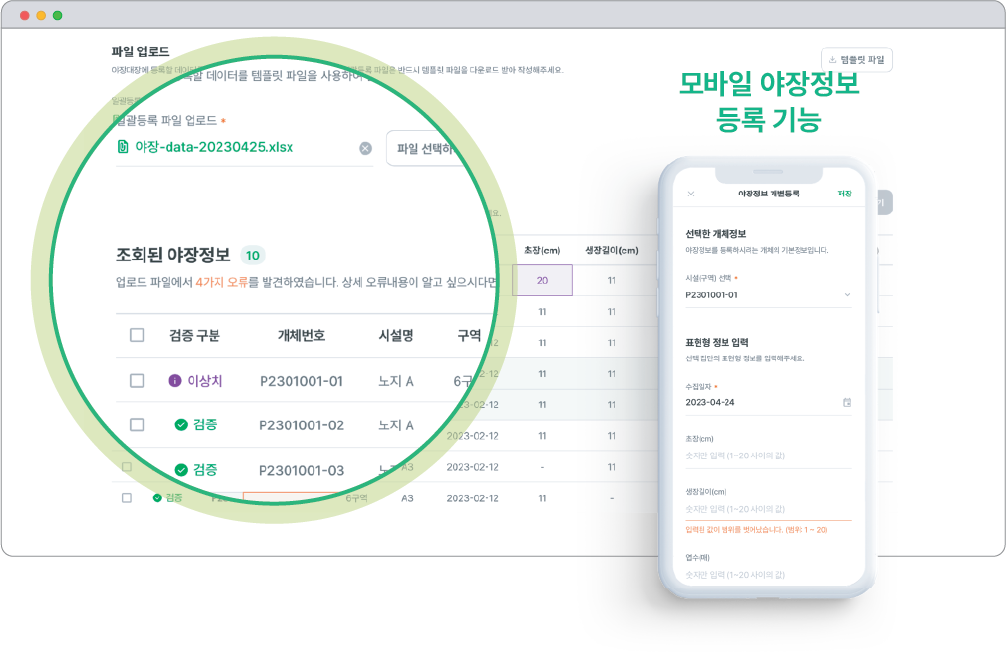
기존에 육성가분들의 수기작성법으로 기록할 때 발생할 수 있는 데이터 통합의 어려움과 검색 및 활용의 한계를 극복하기 위해, 대용량 야장 정보를 규격화하여 관리하는 기능을 제공합니다(그림 4).
모바일로도 현장에서 직접 개별 등록이 가능하고, 템플릿을 사용하면 대용량 야장 정보도 손쉽게 일괄 등록할 수 있습니다. 또한 야장 정보 등록 시 우려되는 오타나 오입력 정보를 식별할 수 있는 자동 오류 검사 기능을 지원하여, 사용자가 쉽게 데이터 오류를 인지하고 정정할 수 있습니다.
질적 및 양적 원예 형질 측정값은 물론, 영상, 재배 정보와 같은 다양한 종류와 형식의 데이터를 포괄적으로 등록하고 관리할 수 있으므로 데이터에 기반해 우수 자원을 객관적으로 선발하는 데 매우 유용하게 활용될 수 있습니다.
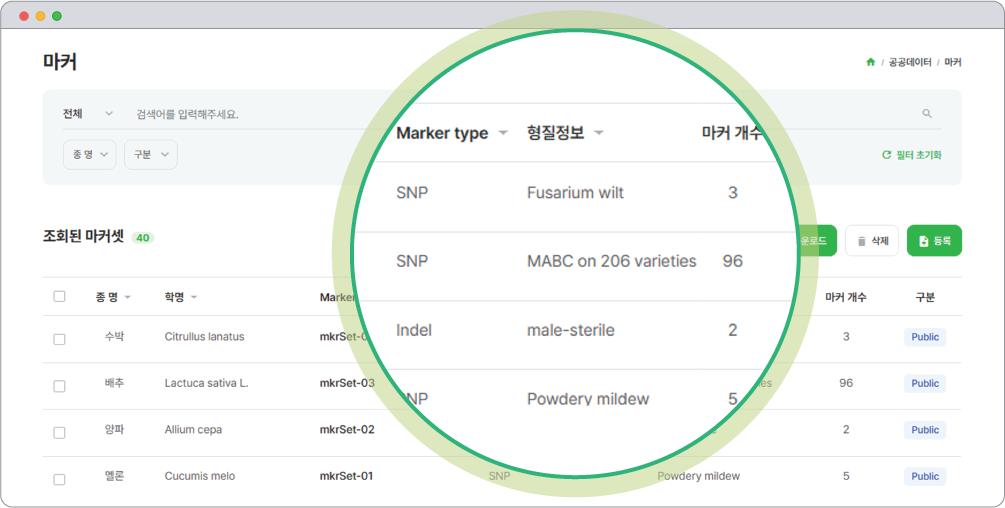
데이터에 근거하여 육종을 위한 객관적인 정보를 도출하기 위해서는 신뢰할 수 있는 데이터가 많으면 많을수록 좋습니다. ibreeder는 유전자원, 유전형, 원예 형질, 마커 등 작물에 대한 모든 데이터를 정규화하여 통합 관리하기 때문에 새로운 데이터가 계속 추가되더라도 이전 데이터와 함께 데이터 육종에 활용될 수 있습니다.
또한 공공데이터(참조 유전체, 마커) 정보를 함께 제공하여 별도의 검색 없이 데이터를 한 곳에서 확인할 수 있어서 손쉽게 접근하고 활용할 수 있으며 유전체 버전이 달라져도 데이터 전환이 용이합니다(그림 5).
어떠한 정보도 소홀히 다루지 않으며, 장기간 보관하고 활용할 수 있도록 데이터의 지속적인 추가와 확장성을 보장함으로써 축적된 데이터를 전통 육종, 분자 육종 그리고 AI 육종(디지털 육종)에 즉각적으로 활용할 수 있습니다. 이를 통해 기후 변화에 대응하고 글로벌 종자 시장에서의 상품성을 강화하는 등 급변하는 육종 목표에 신속하고 효율적으로 대응하는데 이바지할 것으로 기대됩니다.
양파 구중 개량: 시뮬레이션을 통한 교배조합 작성법
본 분석은 2022년부터 2년 동안 국내 양파의 유전형 및 표현형(원예 형질) 정보를 디지털화하고, 기계학습을 활용한 데이터 기반 디지털 육종의 실제 사례를 보여줍니다.
양파 98 계통(계통별 1 개체씩)의 GBS (Genotyping By Sequencing) 분석을 통해 총 51,499개의 SNP를 확인하였고, GEBV method와 ML method를 적용하여 유전체 육종가 및 기계학습 예측 모델을 구축하였습니다.
이를 신규 개체의 실제 유전형을 통해 실측치와 예측치를 교차 비교하여 예측 모델의 정확도를 검증하였습니다. 그리고 해당 모델을 이용해 인실리코 교배시뮬레이션을 수행하여 구중 개량을 위해 우선적으로 교배될 모부본을 제시하고 그 결과를 확인한 것으로, 전체 워크플로는 다음과 같습니다.
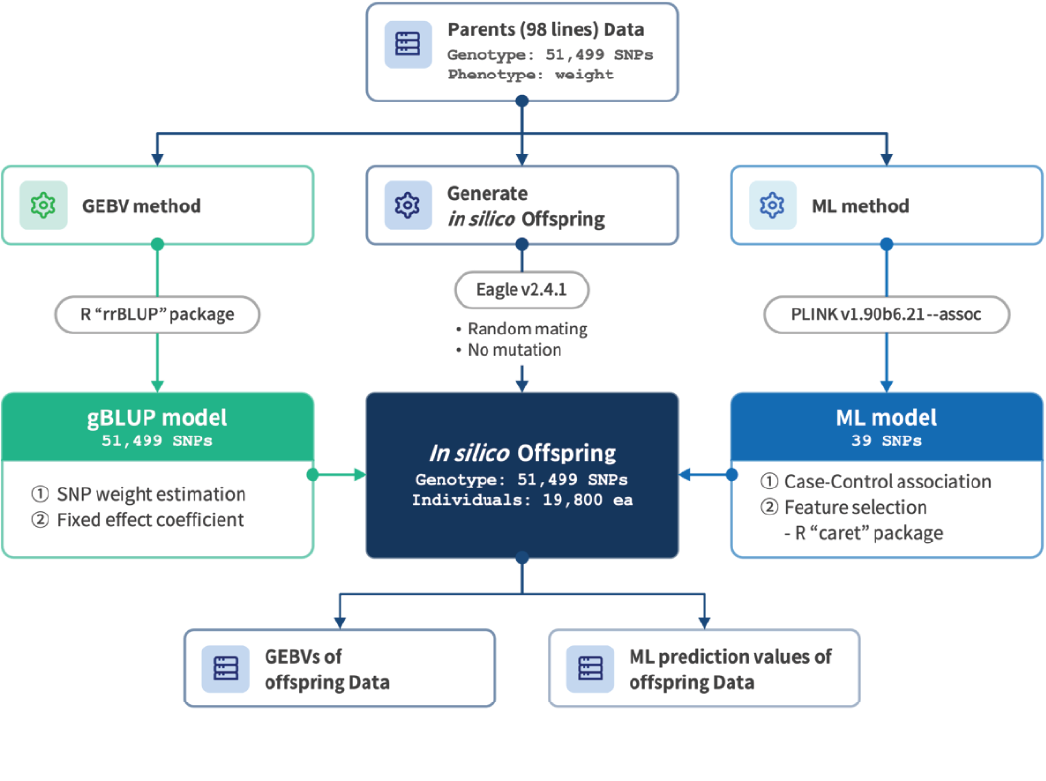
먼저 전체 SNP의 유전적 조성에 따른 구중의 유전체 육종가(Genomic Estimated Breeding Value)를 추정하였습니다(GEBV method).
gBLUP 방법을 이용해 구중에 대한 유전체 육종가를 예측한 결과, 실제값과 예측값 사이에 완벽한 상관관계(r2 = 1.0)가 나타났습니다(그림 7-A). 이 결과는 가계 정보나 재배 환경 데이터 없이 오직 100% 유전형만을 기반으로 예측되었기 때문으로, 일부 과추정 문제로 육종에 직접 활용하기에는 정보가 조금 부족하지만, 인실리코 자손의 표현형을 예측하여 교배조합을 산출하는 데에는 충분히 활용될 수 있습니다.

신규 260 개체의 실제 유전형과 실측치로 모델을 검증한 결과 상관계수( 2)는 0.22로, 평균오차는 106.36g으로 확인되었습니다(그림 7-B). 전체 5만여 개의 SNP 유전형을 확인할 경우 정확도가 훨씬 높아질 수 있지만, 비용과 시간 절감을 위해 39개의 선발된 SNP에 대해서만 분석되었기 때문에 오차가 크게 나타났습니다. 막대그래프에서 확인할 수 있듯이 일부 큰 잔차(예측값과 실제값의 차이)를 제외하고 대부분의 차이는 그리 크지 않기는 하지만, 실제 현장에서 적용 가능할 만큼 적은 개수의 SNP 유전형 분석만으로는 유전체 육종가를 추정하기에 충분하지 않다는 것을 확인할 수 있었습니다(일반적으로 1,000개 이상의 변이를 사용하는 것을 추천하고 있습니다).
두 번째로 유전형-표현형 연관분석(GWAS)을 수행하여 1차 구중 연관 SNP를 1,000개 선별하고 기계학습을 통해 SNP별 구중 예측에 대한 기여도에 따라서 최종 39개 SNP를 선별하여 예측 모델을 구축하였습니다(ML method).

98 계통의 구중 분포를 확인하고 상·하위 30%를 각각 control/case로 간주하여 GWAS 분석을 수행한 결과 통계적으로 유의미한 구중 연관 SNP를 확보하였습니다(그림 8-A, B).
이들 SNP에 대한 계통별 유전형을 토대로 주성분 분석(PCA) 결과 구중과 상관없이 다양한 분포를 보여 구중에 대한 구별력을 확인할 수 없었습니다. 기계학습을 통해 SNP별 구중 예측에 대한 기여도를 기반으로 최종 39개 SNP를 선별했을 때, 첫 번째 주성분을 기준으로 case (파란 점)와 control (빨간 점)이 서로 구별되는 분포의 차이를 보여 구별력이 크게 향상되었음을 확인할 수 있습니다(그림 8-C). 이처럼 기계학습 예측 모델을 구축할 때는 어떤 SNP 마커를 선발하느냐에 따라 정확도가 달라질 수 있으므로, 정확한 마커 정보를 선별하기 위해 GWAS나 문헌 조사가 필수적입니다.
그러나 선별된 마커에도 불구하고, 여전히 구중과 상관없이 서로 혼재된 군집을 확인할 수 있습니다(그림 8-C의 두 번째 PCA plot). 여기에서 기계학습의 필요성을 확인할 수 있습니다. 만약 눈으로 명확하게 구중을 구별할 수 있었다면 소수의 분자 마커나 간단한 회귀식을 통해 충분히 설명할 수 있었겠지만, 여러 유전 인자들이 복잡하게 관여하여 나타나는 양적형질의 경우 기계학습 결과 각 SNP 마커별 가중치를 세밀하게 설정한 수식을 통해서만 정밀한 예측이 가능해지는 것입니다. 즉, 주어진 변이 형태를 다양하게 학습하여 문제를 해결합니다.
실제로 신규 260 개체의 실제 유전형으로 "기계학습 예측 모델"을 검증한 결과, 선별된 39개의 SNP만으로도 구중에 대한 충분한 구별력을 확인했습니다(그림 8-D). 또 AB (AdaBoost)부터 SVM (Support Vector Machine)까지 다양한 7가지 알고리즘을 적용했을 때, 구중을 정확하게 예측한 확률이 83.2%로 나타났는데 이는 10 개체 중 8 개체 이상의 구중을 정확하게 예측할 수 있음을 의미합니다(그림 8-E). 구중의 값(g)을 예측하는 GEBV method와는 달리 기계학습의 경우 구중의 집단 내 경향(상위/하위)을 예측하는 것이기 때문에 적은 수의 SNP 마커만으로도 높은 정확도를 나타낼 수 있습니다. 이처럼 집단 내 엘리트 자원을 선발할 때, 기계학습을 통해 소수의 마커로 우수/불량 여부를 확인하면 저비용으로도 우수 자원 선발의 효율성을 크게 향상할 수 있습니다.
마지막으로 In silico simulation을 통해 98 계통 모부본의 가상 자손(F1)을 생산하고, F1의 구중을 1) GEBV method와 2) ML method를 통해 각각 예측하고, 결과를 비교하였습니다(In silico Offspring).

시뮬레이션을 통해 생산된 19,800개의 인실리코 자손의 유전형이 모부본과 유사한 양상을 보였고, GEBV method를 적용하여 구중을 예측한 결과 모부본의 분포와 동일하게 정규분포를 나타내 데이터의 신뢰성을 확인할 수 있었습니다(그림 9-A).
이때 39개의 선별된 SNP만을 사용했을 때도 유사한 구중 분포가 나타났으나, 전체 51,499개의 SNP에 기반한 예측 결과와는 56.6%의 상관관계만을 보였습니다(그림 9-B). 이는 유전체 육종가를 효과적으로 추정하기 위해서 수만 개의 SNP 유전형 정보가 필요하다는 점을 시사하며, 이러한 대규모 SNP 분석은 실험 비용과 실제 육종을 위한 노동력 측면에서 어려움을 야기합니다.
반면, 기계학습 예측 모델과 교배조합 시뮬레이션을 활용한 결과에서는 우수한 상위 5%와 미흡한 하위 5%의 자손들의 모부본이 실제 엘리트 계통 내 각 상위 하위 5% 안에 포함되는 개체들로 확인되었습니다(그림 9-C, Random forest 알고리즘 기준 구중 예측값 상/하위 5%에 해당하는 자손의 유전형이 구중에 따라 명확하게 구별되는 특성을 보였으며, 교배조합에 사용된 모부본의 구중 역시 같은 분포를 보이고 있음을 확인함). 해당 결과는 그간 육성가분들에 의해 진행되던 표현형 기준의 선발 및 교배조합 작성과 일치하는 것으로 표현형 관찰을 위한 오랜 시간 대신 유전형으로 조기에 선발하고 시뮬레이션을 통해 확률 높은 소수의 교배 조합만을 실천할 수 있도록 하는 효율적인 데이터 육종의 가능성을 보여주었습니다.
양파 98 계통의 모든 개체별 유전형과 표현형 정보를 토대로 생물정보 분석을 통해서 원예 형질(구중)을 예측하고, 신규 개체를 통해 예측 모델의 정확도를 검증하며 교배조합 시뮬레이션을 통해 우수 교배조합을 선발하는 일련의 과정을 확인해 보았습니다. 현재 교배조합 시뮬레이션 결과 선발된 교배조합별 자손의 구중을 검증하기 위해 실험을 진행하고 있습니다. 특히 양파의 경우 2년에 1세대가 진전되어 육종에 오랜 시간이 소요되는 작물이므로, 본 연구의 성과로 세대를 크게 단축함으로써 신속하고 효율적인 육종이 가능할 것으로 기대됩니다.
고추 여교잡 육종: MABC 마커 탐색
MABC (Marker-Assisted Backcrossing) 마커는 여교잡 육종에서 공여친의 관심 유전자 좌위를 제외한 나머지 유전적 특성을 모두 반복친으로부터 물려받은 자손을 선발하는 데 활용됩니다. 따라서 모부본의 유전적 양상을 유전체 전반에서 확인하고, 여교잡 자손에서 유전형 분석을 통해 모부본의 DNA를 추적할 수 있는 MABC 마커를 개발하는 것이 핵심입니다. 본 연구에서는 고추 80 계통의 WGS (Whole Genome Sequencing)를 통해 고품질의 2,997,866 SNP을 확보하고, 80 계통을 모부본으로 활용 시 여교잡 1~2세대 안에 선발할 수 있는 마커를 선별하였습니다(그림 10).

분석에 활용된 80 계통은 신미, 과장, 과형 등 6개의 주요 원예 형질에 따라 나뉘며, 주성분 분석을 통해 실제로 각 계통의 유전적 양상 역시 원예 형질에 따라 명확하게 구별됨을 확인할 수 있었습니다(그림 11-A). 계통별 구별(추적)이 가능한 마커를 선별하기 위해서 모든 SNP 유전형을 비교하여 마커를 선별하였습니다. 선별된 SNP 마커가 초기 의도한 대로 고추 유전체 전반에 걸쳐 모든 염색체에 고르게 분포하는 것을 확인하였으며(그림 11-B), heatmap을 통해 계통별 다형성을 확인할 수 있었습니다(그림 11-C).
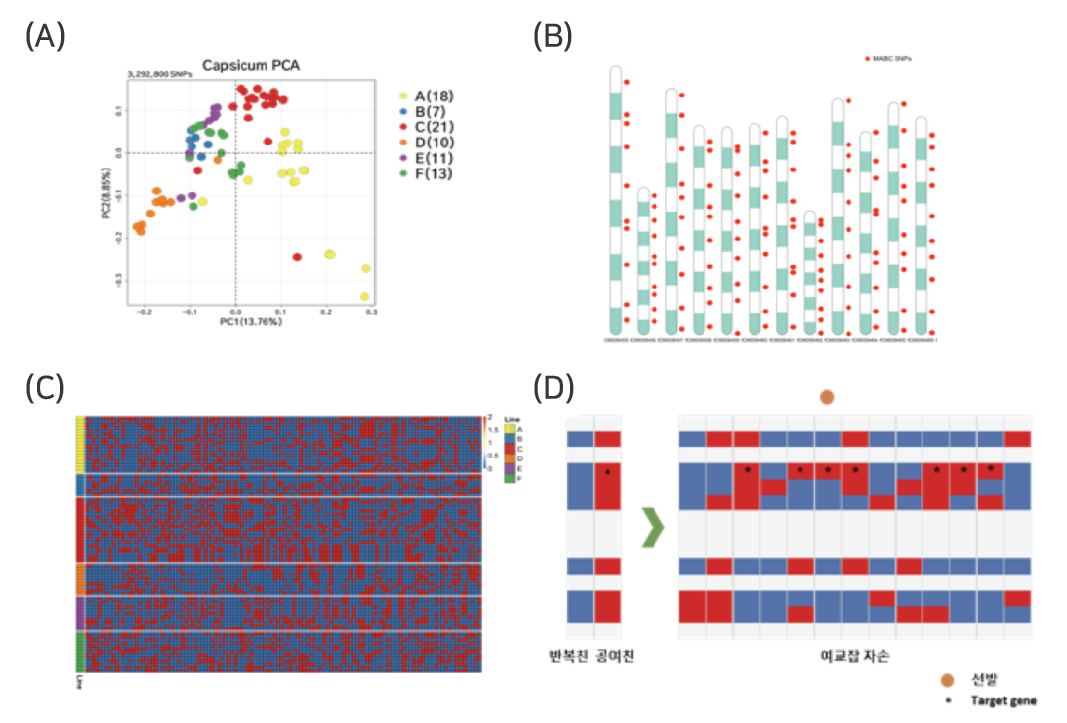
그 결과, 선별된 108개의 마커를 적용하면 전체 80 계통이 각각 반복친으로 사용되어도 여교잡 자손의 background 선발이 가능합니다. 특히 주요 25 계통에 대해서는 단 12개의 마커만으로도 여교잡 자손을 효율적으로 선발할 수 있습니다.
MABC 마커는 다음과 같이 TaqMan, KASP, rhAmp SNP 등 다양한 실험 방법에 따라 적용하실 수 있습니다(그림 12).

도입하고자 하는 신미, 병 저항성 등 목표 원예 형질에 대해 기계학습 기반 예측 결과 foreground 선발을 수행하고, MABC 마커를 적용하여 background 선발을 진행하면 여교잡 1~2세대 만으로도 계통 육성이 가능합니다. 이 방법은 분자 육종과 디지털 육종을 결합한 데이터 육종으로 근본적인 육종 과정의 효율성을 크게 증진시켜 빠르고 정확한 품종 개발을 도울 수 있습니다.
분자 마커 실험·검증 GUI 솔루션 : CLC Main Workbench
CLC Main Workbench는 프로그래밍이나 리눅스 환경에 대한 기초 지식이 없어도 생물정보 데이터를 손쉽게 처리할 수 있는 GUI 환경의 분석 소프트웨어입니다. 연구자들이 육종을 위한 생물정보 데이터를 분석할 때 쉽게 확인하고, 복잡한 분석을 직접 간단한 작업으로 수행할 수 있게 도와줍니다.
MABC, 웅성불임, 품종판별, 마커 이용 선발(MAS) 등 다양한 분자 마커의 실험을 위한 프라이머 서열을 간편하게 디자인할 수 있습니다. 번거롭게 서열을 준비하고 다형성 부위를 별도 표시할 필요 없이 원하는 영역을 드래그하여 선택하고, 몇 번 간단히 클릭만 해도 프라이머 목록을 확인할 수 있습니다. 이때 HRM, KASP, CAPS, SCAR, TaqMan 등 다양한 유형의 프라이머를 디자인할 수 있도록 파라미터를 설정할 수 있으며, Standard PCR, TaqMan 등 주요 유형의 경우 기본 옵션으로 제공되어 간단히 적용할 수 있습니다. 또한 주어진 프라이머의 GC contents, product size, temperature 등 주요 특성을 토대로 실험에 적합한 프라이머를 쉽게 선정할 수 있습니다(그림 13).
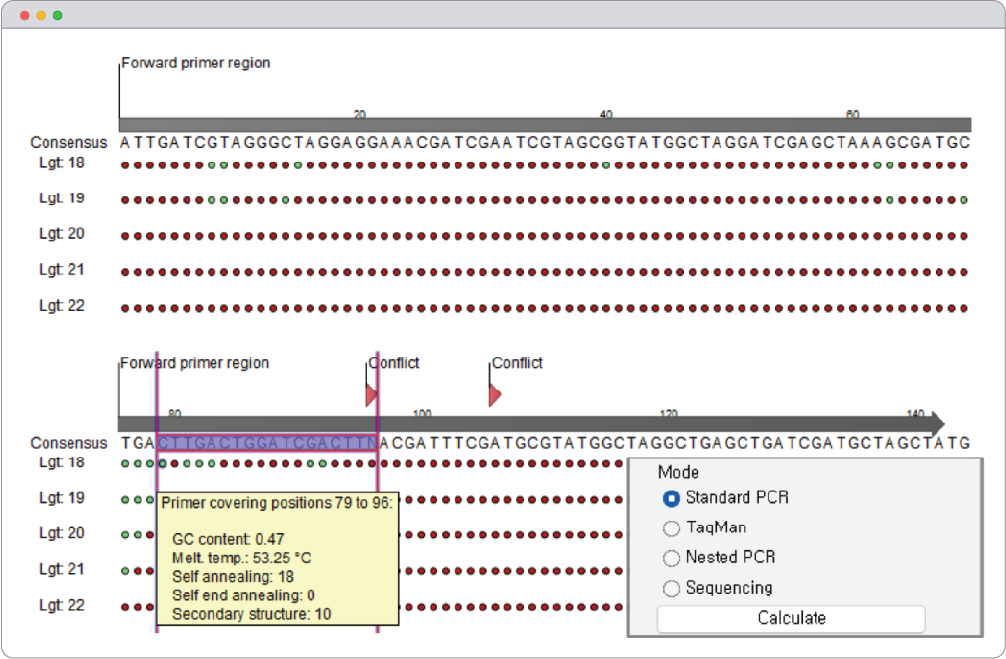
프라이머를 통해 유전형을 분석한 후, 실제로 계통별 어떤 다형성을 보이는지 확인하는 것이 필요합니다. CLC Main Workbench는 관심 영역에서 계통별 유전형 실험 결과를 직접 확인할 수 있도록 시각화 기능을 제공합니다. 다중서열정렬(Multiple sequence alignment) 기능을 이용하여 병저항성/감수성 자원 간 Indel 영역을 비교해 본 결과를 예시로 들 수 있습니다(그림 14). 프라이머에 의해 증폭되는 영역과 감수성 자원에서의 deletion 영역을 한눈에 확인할 수 있어 용이하며, 약간의 가공만으로도 논문이나 보고서의 그림으로 사용할 수 있는 장점이 있습니다.

또한 계통 간 유전적 진화 유연관계를 파악하고자 할 때, 계통분석 기능을 활용할 수 있습니다. 병 저항성 등 자원의 관심 원예 형질 특성을 계통수 내에서 함께 확인할 수 있어 유전적 다양성을 고려한 유용 자원 선발에 용이합니다(그림 15).

현재 연구의 디지털 전환을 위한 프로모션의 일환으로, CLC Main Workbench의 1년 라이선스 무료 제공 이벤트가 진행 중입니다.
또한 ㈜인실리코젠의 온라인 교육 사이트 incoEDU를 통해 제공되는 평생 무료 강의를 통해 쉽게 배울 수 있으니, 이참에 자신의 데이터를 직접 입맛에 맞게 분석하여 육종에 활용해 보시기 바랍니다.
마치며
구슬이 서 말이라도 꿰어야 보배라는 말이 있듯이 다양하고 예쁜 디자인의 주얼리(신품종)를 제작(분자 육종, 디지털 육종, 분자 마커 실험·검증 솔루션)하려면, 먼저 구슬을 모으는 과정이 선행되어야 합니다(ibreeder).
병 저항성, 시장 선호도 등 기존 목표뿐만 아니라 K종자의 세계화, 기후변화에 따른 가뭄 저항성, 스마트팜에 적합한 작물 육성까지 글로벌시대 식물육종의 새로운 변화에 효과적으로 대응할 수 있도록 데이터를 모으고 분석할 필요가 있습니다.
데이터의 가치를 잘 알고, 생물정보 분석 노하우를 축적한 (주)인실리코젠이 데이터로부터 시작되는 지속 가능한 농업을 실현할 수 있도록 열심히 노력하겠습니다.
함께 읽어보면 좋은 글

EDITOR
정명희
R&D Center · Deputy Principal Researcher
'STORAGE' 카테고리의 다른 글
| 사이버 보안의 중요성: 보안 위협 유형 (0) | 2024.07.18 |
|---|---|
| [Lab Story] 성균관대학교 윤환수 교수님 편 (0) | 2024.07.08 |
| 쯔쯔가무시병에 대해 아시나요? (0) | 2024.06.10 |
| 인실리코젠 프로젝트: 데이터 육종 I (0) | 2024.05.27 |
| 건강기능식품, 효과만큼 주의사항도 체크하세요! (0) | 2024.05.09 |






댓글