
인실리코젠이 Lab Story 제2편으로 돌아왔습니다~
이번에는 중앙대 안성 캠퍼스의 김준모 교수님을 만나 뵙고 왔는데요.
김준모 교수님은 2013년부터 중앙대 생명공학대학 생명자원공학부 동물생명공학전공 학생들을 지도하고 계십니다.
주요 연구 분야로는 생물정보학을 이용한 다양한 동물유전현상의 이해, 복합유전체정보의 해석, SNP Chip 개발과 동물의 기능성 유전자 발굴, 축산분야 빅데이터를 활용한 생물정보 기술 개발 등이 있습니다.
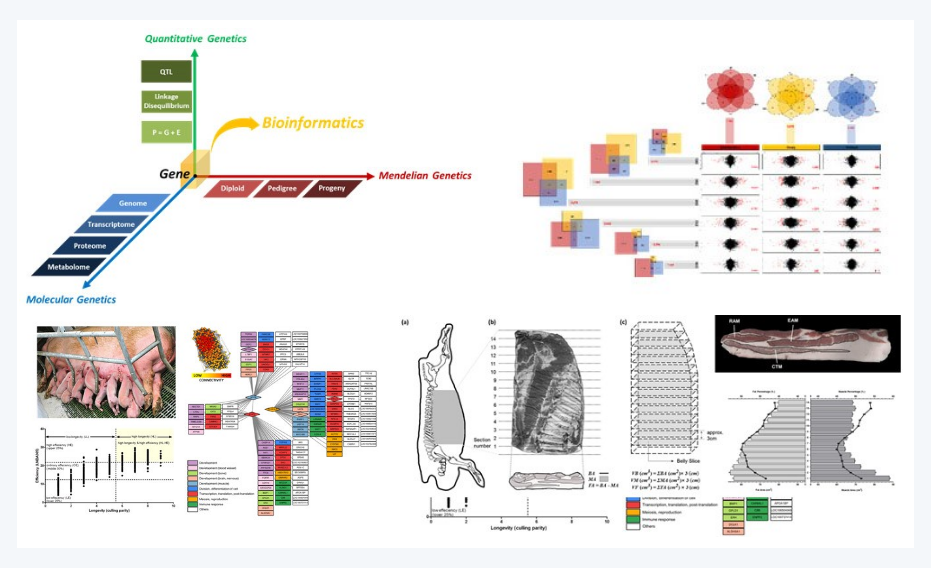
Dry Lab으로서 많은 데이터를 가지고 연구를 진행한다는 점이 저희 인실리코젠과 닮아 있는데요! 여러 동물의 유전체 데이터를 다루며 기능유전체 분석법을 이용하여 질병, 약리 활성, 영양, 동물복지 등 축산분야에 적용하고 계신 김준모 교수님의 BIGKIM LAB으로 가 보시죠!
Introducing the Professor
Q. 人Co 블로그 구독자 분들께 자기소개와 어떤 연구를 중점적으로 하고 계신지 말씀 부탁드립니다.

안녕하세요, 중앙대 생명공학대학 동물생명공학전공 김준모 교수입니다.
저희 기능유전체·생물정보 연구실에서는 각종 유전체 데이터를 다루며, 주로 멀티오믹스통합(MOI) 분석기법을 활용해서 생리대사 기전 및 타겟을 발굴하는 연구를 진행하고 있습니다.
주 연구로는 면역유전체학 연구, 유전자 발현량 기반의 통합분석 네트워크 연구, 동물 유전체와 미생물 유전체 간의 상호작용 연구, mRNA & IncRNA 연관성 분석, 전장 유전체 기반의 질병 역학 연구, 후성유전학 연구 등을 진행하고 있습니다.
대표적으로 2016년에 진행된 돼지-영장류 이종 장기 이식에 대한 전사체 통합분석 기반 이식거부반응 및 부작용 매커니즘 규명을 통해 거부반응조절제 개발에 참여했습니다.

세계적으로 ‘이종 간 장기이식’ 연구가 활발히 진행되고 있는데요, 이종 장기 이식은 유전적 변형 유무와 관계없이 동물의 살아있는 장기나 조직, 세포를 사람에게 이식하는 것을 의미합니다.
국내에서는 면역거부반응을 제어한 돼지의 심장을 원숭이에게 넣은 뒤, 51일 동안 건강하게 생존하는 데 성공하였고 저희 BIGKIM LAB에서는 이 과정에 필요한 거부반응조절제 연구에 참여하였습니다.
Introducing the Lab
Q. 열심히 연구하고 계신 연구원분들이 많아 보여요! 현재 연구실의 멤버 구성은 어떻게 되나요?

현재 2명의 포스트닥 박사님들과 6명의 박사 과정 학생, 2명의 석사 과정 학생, 그리고 3명의 학부 연구생이
함께하고 있습니다.
Q. 연구실이 일반적인 연구실과 조금 다르게 느껴지는데요. 또 연구실 곳곳에 BIGKIM 로드, BIGKIM 어워드 등 BIGKIM이라는 키워드가 자주 보이는 것 같아요. 이에 대해 말씀해주실 수 있을까요?

저희 연구실은 서버실을 중심으로 책상 위 컴퓨터와 사람만 존재하는 Dry Lab 공간으로, 흔히 연상되는 하얀 가운과 파이펫을 잡고 실험하는 연구실과는 분위기가 많이 다른데요. 이 점이 여타의 다른 연구실과 차이점이라면 가장 큰 차이점입니다.
BIGKIM은 저희 연구실의 가장 소중한 자산인 HPC(High Computing Performance) 서버의 이름이 BIGKIM이에요. 제 성이 김씨라서 그렇게 지은 거라 별 뜻은 없습니다.

Dry Lab이란?
Dry Lab이란 연구자가 실제 실험환경이 아닌, 가상의 실험환경에서 연구한다는 의미에서 부르는 용어인데요, 컴퓨터에서 신뢰성 있는 데이터를 기반으로 다양한 실험을 한다는 점에서 in Silico Lab이라고 부르기도 합니다. Dry Lab에서 핵심 도구는 바로 연구 데이터라고 할 수 있습니다. 반면 실제 실험환경은 수많은 실험 장비와 시료를 포함하며 Wet Lab 또는 in Vitro Lab이라고 불린답니다.
Q. 기능유전체, 생물정보 연구실을 한 문장으로 표현한다면 어떻게 얘기할 수 있을까요?
한 문장도 필요 없겠네요. “GOATs(Greatest of All Time)” 최고의 인물들이 모여 있는 곳이라고 생각합니다.
Introducing the Lab
Q. 최고의 인물들이 모인 연구실은 일하는 방식이나 문화도 다를 것 같은데요. 연구실만의 특징이 있다면 소개해주세요.

저희 연구실에서 가장 소중한 자산이 HPC 서버라고 소개해드렸는데요. HPC 서버는 병렬방식의 연결구조로 컴퓨팅 파워를 높여주게 됩니다. 저희 연구실도 수평구조의 연구방식을 지향해 서로가 서로에게 시너지 효과를 내고, 이를 기반으로 효율적이고 능동적인, 그리고 창의적인 연구를 진행하고 있습니다.
Q. 다른 기관이나 기업과 함께 연구를 진행할 때도 있으실 것 같은데요. 인실리코젠과는 어떻게 인연을 맺게 되셨나요?
인실리코젠에 ‘인’을 사람 인(人)자를 쓰더군요. 인실리코젠의 사람과 아주 오래된 인연으로 시작해 인실리코젠과 인연을 맺게 된 것 같네요.
Q. 연구실에 관심이 있는 학생들이 참고하면 좋을 연구실의 대표적인 논문은 무엇이 있을까요? 추천 부탁드립니다.
더 많은 논문은 https://www.bigkim.org/ Journal Club과 Publication 탭 참고 바랍니다.
- Marinus te Pas, Woncheoul Park, Krishnamoorthy Srikanth, Himansu Kumar, Steve Kemp, Jun-Mo Kim, Dajeong Lim, Ole Madsen, Henry van den Brand, Jong-Eun Park (2023) Transcriptomic and epigenomic network analysis reveals chicken physiological reactions against heat stress. Transcriptome Profiling. Page 333-359. (BOOK CHAPTER). (https://doi.org/10.1016/B978-0-323-91810-7.00002-9)
- Rajesh Kumar Pathak and Jun-Mo Kim (2023) Targeting PEDV 3CL protease for identification of small molecule inhibitors: An insight from virtual screening, ADMET prediction, molecular dynamics, free energy landscape and binding energy calculation. Journal of Biological Engineering. Accepted.
- Seung-Hoon Lee and Jun-Mo Kim (2023) Genetic parameters of the pork belly indicate correlation of genetic traits of belly and carcass with meat quality. Animal bioscience. Accepted.
- Seung-Hoon Lee and Jun-Mo Kim (2023) Breeding potential for pork belly to the novel economic trait. Journal of Animal Science and Technology. 65(1):1-15. (https://doi.org/10.5187/jast.2022.e118)
- Yoon-Been Park and Jun-Mo Kim (2022) Identification of lncRNA-mRNA interactions and genome-wide lncRNA annotation in animal transcriptome profiling. Journal of Animal Science and Technology. Accepted. (https://doi.org/10.5187/jast.2023.e17)
- Seung-Hoon Lee, Sang-Hoon Lee, Hee-Bok Park, and Jun-Mo Kim. (2022) Identification of Key Adipogenic Transcription Factors for the Pork Belly Parameters via the Association Weight Matrix. Meat Sci. 195:109015. (https://doi.org/10.1016/j.meatsci.2022.109015)
- Gi Ppeum Han, J. H. Kim, Jun-Mo Kim and Dong Yong Kil (2022) Transcriptomic analysis of the liver in aged laying hens with different eggshell strength. Poultry Science. 102:102217. (https://doi.org/10.1016/j.psj.2022.102217)
- Yejee Park, Min-Jae Jang and Jun-Mo Kim (2022) Transcriptome profiling and genomic atlas study for endocrine disrupting chemicals in whole body system of animals. Journal of Animal Breeding and Genomics. 6(4)195-200. (https://doi.org/10.12972/jabng.20220021)
- Jun-Mo Kim (2022) Genetic parameters of muscle fiber traits and association of a novel polymorphism BspCNI MYOG locus in pigs. Journal of Animal Breeding and Genomics. 6(4)173-181. (https://doi.org/10.12972/jabng.20220019)
- Rajesh Kumar Pathak and Jun-Mo Kim (2022). Vetinformatics from functional genomics to drug discovery: Insights into decoding complex molecular mechanisms of livestock systems in veterinary science. Frontiers in Veterinary Science. 9:1008728. (https://doi.org/10.3389/fvets.2022.1008728)
- Do-Young Kim, Kyung-Tai Lee, Eun-Seok Cho, Woncheol Park, Kyu-Sang Lim, Si Nae Cheon, Bong-Hwan Choi, Dajeong Lim, Jong-Eun Park, and Jun-Mo Kim (2022) Integrative Methylome and Transcriptome analysis of Porcine Abdominal Fat indicates Changes of Fat Metabolism and Immune Responses during Growing and Finishing Periods. Journal of Animal Science. 100(12). (https://doi.org/10.1093/jas/skac302)
- Chiwoong Lim, Byeonghwi Lim, Dong Yong Kil and Jun-Mo Kim (2022) Hepatic transcriptome profiling according to growth rate reveals acclimation in metabolic regulatory mechanisms to cyclic heat stress in broiler chickens. Poultry Science. 102167. (https://doi.org/10.1016/j.psj.2022.102167)
Q. BIGKIM LAB만의 복지가 있다면?

BIGKIM LAB만의 복지라고 한다면, 별건 아니지만 연구하는 학생들이 먹을 걱정은 하지 않도록 다양한 종류의 음료와 컵라면을 제공하고 있습니다. 아무래도 연구에 몰두하거나 집중하게 되면 끼니를 거를 때가 많은데, 간단하게라도 식사를 해결할 수 있게 하고 있습니다. 그렇다고 밤낮없이 연구만 하는 건 아니랍니다 😊
Be Inspired!
Q. 연구실의 랩장을 맡고 계신 임치웅 선생님께 기능유전체, 생물정보 연구실에 대해 학생의 입장에서 이야기를 들어보고 싶은데요. 대학원 과정으로 기능유전체, 생물정보 연구실을 선택하게 된 계기는 무엇인가요?

3학년 2학기 때 동물유전체학, 동물육종학및생체정보학, 동물분자세포생물학 등 생물정보학의 기반이 되는 강의를 수강했는데요. 이러한 과목을 공부하면서 진로에 대해 고민을 하던 중 transcriptome을 기반으로 분석을 진행하는 RNA-sequencing에 대해 관심을 가지게 되었습니다.
이를 계기로 3학년 겨울방학에 기능유전체, 생물정보 연구실에 인턴으로 지원하여 생물정보학의 기초적인 배경과 지식을 배울 수 있었고, 분석에 이용되는 통계, 분석, 시각화를 위한 프로그램들을 다루는 법을 배우면서 가공한 데이터의 결과를 분석하고 표현하기 위해 작성하는 코드에도 집중해 볼 수 있었습니다. 이때, 분석에 필요한 코드를 배우고 이를 적용해 RNA-sequencing 과정을 수행하는 것에 큰 흥미를 느끼게 되었습니다.
기능 분석을 위해 필요한 지식에 대한 자료 조사를 하고 공부하는 과정에 더 공부를 해보고자 졸업 후 기능유전체, 생물정보 연구실에 지원하고 함께하게 되었습니다.
Q. 분석에 필요한 코드를 배우고 이를 적용해 RNA-sequencing 과정을 수행하는 것에 큰 흥미를 느끼셨다고 하셨는데 연구를 진행하며 가장 기억에 남는 프로젝트가 있을까요? 어떤 역할을 했는지 간단한 설명해 주세요.
연구를 진행하며 가장 기억에 남는 프로젝트는 아무래도 제 첫 논문이었던 돼지의 등 지방 전사체 데이터 자료를 확인하여 성장에 따른 유전자 발현의 변화를 확인한 연구였습니다.
연구를 진행하면서 이론으로만 배웠던 분석과정에 대해서 직접 부딪히고 빈틈을 메꾸면서 앞으로의 연구 생활에 있어서 꼭 필요한 능력을 기를 수 있었거든요. 그 과정에서 연구의 기쁨을 깨달을 수 있는 시간을 보냈던 것 같아요.
또한 논문을 작성하는 과정에서도 연구자의 필수 역량이라고 할 수 있는 연구논문 작성 과정에 대한 첫 경험을 겪음으로써 앞으로의 연구자 인생에 첫발을 내딛는 경험을 하게 되어 가장 크게 기억에 남는 프로젝트였다고 생각됩니다.
Q. 기능유전체, 생물정보 연구실(또는 타 대학원)을 고민하는 학생들에게 해줄 수 있는 조언이 있나요?
실험을 주로 하는 다른 연구실과는 다르게 생물정보분석을 진행하는 연구실의 경우 주로 dry lab인 경우가 많습니다. 우리 연구실도 마찬가지로 dry lab인데 그 특성 상 직접 몸을 움직여 실험을 진행하는 것이 아닌 컴퓨터를 이용해 분석을 진행하고 연구하여 더 생소하게 느껴질 수도 있을 것으로 생각됩니다.
하지만, 연구 분야의 특성에 따라 동물, 식물, 인간 어떤 것이든 연구에 적용할 수 있으며 다양한 연구 분야에 관한 포괄적인 연구가 가능하다는 장점이 존재합니다. 최근 거의 모든 과학적인 연구 분야에 생물정보학을 필요로 하는 만큼 이 연구 분야의 중요성이 부각되며 연구실 혹은 기업 인턴 등을 통해 생물정보학 및 분석에 대한 여러 경험을 바탕으로 한다면 큰 도움이 될 것 같다고 생각합니다.

Reference
- http://www.rda.go.kr/board/board.do?mode=view&prgId=day_farmprmninfoEntry&dataNo=100000727577
- https://dataonair.or.kr/db-tech-reference/d-story/data-interview/?mod=document&uid=63320
- https://www.bigkim.org/
Lab Story 1편을 보고 싶다면? 👇👇👇
>> [Lab Story] 충북대학교 조성진 교수님 편
인실리코젠의 디지털 마케터가 전하는 생물정보 이야기

EDITOR
Digital Marketer
FLEX Dept.
'STORAGE' 카테고리의 다른 글
| 잠은 죽어서 자라? 아니죠! 당신이 잠을 잘 자야 하는 4가지 이유 (0) | 2023.06.19 |
|---|---|
| 손쉽게 웹사이트 검색 엔진 최적화(SEO)하는 5가지 방법 (0) | 2023.06.05 |
| 탈모에 대한 속설, 과연 사실일까? (0) | 2023.05.08 |
| "ChatGPT"가 뭐길래?! (0) | 2023.04.10 |
| [Lab Story] 충북대학교 조성진 교수님 편 (0) | 2023.03.27 |






댓글