
NGS 데이터는 처리하는 과정에서 다양한 형식의 파일이 생성됩니다. 그리고 각각의 분석 과정에서 지정된 데이터 포맷의 파일만 입력 파일로 활용할 수 있고 분석 결과는 지정된 포맷의 파일이 생성됩니다.

예를 들면 위의 분석 과정 중 alignment to genome 단계에서 자주 사용되는 bwa 이라는 분석 툴은 FASTQ 형식 파일을 입력 파일로 받아서 SAM 형식의 결과 파일을 출력합니다. 이렇게 분석 단계별로 입출력 파일 형식이 지정되어 있습니다.
대용량의 NGS 데이터를 분석하다 보면 예기치 못한 오류메시지를 보게 되는 경우가 종종 있습니다. 이런 때 의외로 데이터 파일 포맷이 올바르지 않아 프로그램에서 인식할 수 없다는 로그를 종종 보게 됩니다.
원인은 다양합니다. 우선 연구자가 임의로 만든 파일이 지정된 형식을 지키지 않은 경우가 있겠지요. 또는 시퀀싱 오류, 파일 전송 중이나 분석 과정 중 오류가 발생한 경우 등 여러 가지 원인에 의한 파일 손상 때문일 수도 있습니다.
분석 중에 파일 포맷과 관련된 에러 메시지를 보고 파일의 어느 부분에 문제가 있는지 확인해야 하는 경우가 종종 있습니다. 우연히 파일 첫 번째 줄에서 문제를 발견할 수도 있고, 검토할 파일 크기가 작아서 문제가 되는 부분을 쉽게 찾을 수 있는 상황이라거나 파일 용량이 크긴 하지만 시간이 많아서 장인의 마음으로 한 줄 한 줄 읽어서 확인할 수 있는 상황이라면 큰 문제가 되지 않습니다. 하지만 대부분 작게는 수백 메가바이트(MB)에서 수 기가바이트(GB) 단위의 여러 개의 파일을 다루어야 하고 사람이 읽을 수 없는 바이너리 파일인 경우도 있습니다. 무엇보다 현대인의 하루는 몹시 바쁜 법이죠. 이번에는 몹시 사소하지만, 알고 있으면 가끔 편리하게 활용할 수 있는 NGS 데이터 유형별 파일 포맷을 검증을 도와줄 수 있는 리눅스 기반의 분석 툴과 사용 방법을 간단하게 소개해 드릴까 합니다.
FASTQ 파일
서열 정보를 포함한 파일입니다. 네 개의 줄이 하나의 서열 정보를 나타냅니다. FASTQ 파일의 형식은 아래와 같습니다.

FASTQ 파일의 각 라인에 대한 상세한 설명이 궁금하신 분들은 여기로 가시면 상세한 설명을 보실 수 있습니다. FASTQ 파일 포맷을 검증이 필요할 때 ValidateFastQ 툴을 사용할 수 있습니다. 2022년 11월 기준 0.1.1 버전을 제공하고 있습니다. FASTQ 파일의 포맷을 검증해주며 중복되는 서열 데이터 여부, ASCII 코드로 표현되는 각 서열의 품질 점수가 생성되는 시퀀서 플랫폼에 따라 유효한 문자인지를 검증해줍니다. Paired-end 서열 데이터 두 파일의 reads 들이 같은 헤더 정보를 가지는지, 두 파일의 서열 수가 같은지 등을 검증해줍니다. JAR 파일로 구성되어 있어 JAVA 애플리케이션을 구동할 수 있도록 JRE 등을 설치 후 커맨드 라인으로 명령어를 입력하여 사용할 수 있습니다. 리눅스 운영체제에서 JRE 설치 방법은 이곳을 참조하여 설치하실 수 있습니다. ValidateFastQ는 여기서 다운로드할 수 있습니다. 전체 분석 옵션 목록 및 상세 설명 등은 여기에 설명되어 있습니다. 사용 방법은 아래와 같이 검증할 FASTQ 파일 경로를 입력해 주면 됩니다.
# ValidateFastQ 사용 방법 (Single end 인 경우) $ java -jar validatefastq-assembly-0.1.1.jar -i <FASTQ file> # ValidateFastQ 사용 방법 (Paired end 인 경우) $ java -jar validatefastq-assembly-0.1.1.jar -i <R1 FASTQ file> -j <R2 FASTQ file>
아래는 정상적인 형식의 FASTQ 파일을 입력 파일로 지정하여 실행 예제입니다. 정상적인 파일은 아래와 같은 로그를 출력해 줍니다.
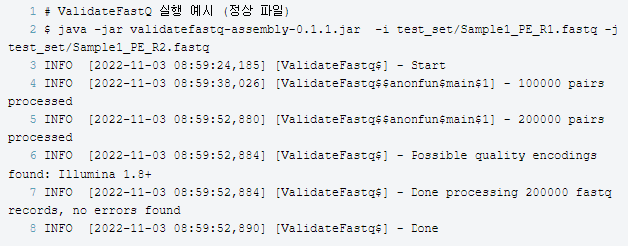
아래는 비정상적인 형식의 FASTQ 파일을 입력 파일로 지정하여 실행 예제입니다. 테스트를 위해 두 개의 파일 중 Sample1_PE_R2.Error.fastq 파일의 서열 데이터를 임의로 삭제 후 입력 파일로 설정했습니다. 아래 로그를 보시면 네 번째 줄에서 파일 포맷에 문제가 있다는 Error found at ... 라는 문구를 확인할 수 있습니다. 다섯 번째 줄에서는 각 FASTQ 파일의 어떤 read 데이터에 문제가 있는지 알려줍니다.

BAM/SAM 파일
BAM/SAM 파일은 FASTQ 파일의 서열 데이터를 참조서열에 정렬하는 과정을 거쳐 생성되는 파일입니다. 각각의 서열 데이터가 참조서열 어느 영역에 정렬되었는지, 해당 서열이 참조서열에 어떻게 정렬되었는지 등에 대한 다양한 정보가 포함되어 있습니다. BAM 파일은 바이너리 파일로 사람은 읽을 수 없는 파일입니다. 이를 사람이 읽을 수 있는 텍스트로 변환된 파일이 SAM 파일입니다. SAM 파일의 형식은 아래와 같습니다.
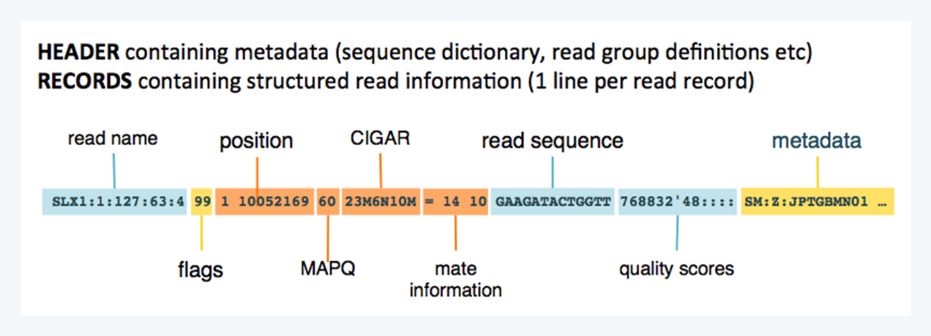
SAM 파일에 대한 자세한 정보는 여기에서 보실 수 있습니다. BAM 파일 포맷을 검증하는 분석 툴로 picard라는 분석 툴의 ValidateSamFile 이라는 모듈이 있습니다. Picard는 2022년 11월 기준 2.27.5 버전을 제공하고 있습니다. BAM/SAM 파일의 형식, 잘못된 서열 정렬 여부, 유효한 플래그(서열 맵핑 정보) 값 여부 등을 검증합니다. JAR 파일로 구성되어 있어 JAVA 애플리케이션을 구동할 수 있도록 JRE 등을 설치 후 커맨드 라인으로 명령어를 입력하여 사용할 수 있습니다. Picard 는 여기서 다운로드할 수 있습니다. 분석 옵션 목록과 상세 설명은 여기를 보시면 상세하게 설명되어 있습니다.
사용 방법은 아래와 같이 검증할 BAM/SAM 파일 경로와 로그 출력 방법을 지정합니다. MODE 옵션을 통해 출력되는 로그 형식을 지정할 수 있습니다.
# picard ValidateSamFile 사용 방법 $ java -jar picard.jar ValidateSamFile -I <BAM OR SAM File> -MODE <SUMMARY OR ERROR OR VERBOSE>
아래는 실행 예제입니다. 아래는 정상적인 형식의 BAM 파일을 입력한 경우 출력되는 로그입니다.

아래는 비정상적인 형식의 BAM 파일을 입력한 경우입니다. 에러 로그 출력을 보기 위해 서열 정렬 툴을 이용해 정렬 분석 수행 중 임의로 프로세스를 중단해서 만든 손상된 BAM 파일을 입력 파일로 지정했습니다. 실행할 때, MODE 옵션을 SUMMARY로 설정해서 오류 유형별 데이터 건수로 간단하게 표기하도록 하겠습니다. 하단의 로그를 보시면 아홉 번째 줄부터 열한 번째 줄까지 입력 파일에서 발견된 오류 유형과 해당 유형에 대한 데이터 수를 요약하여 출력해줍니다.
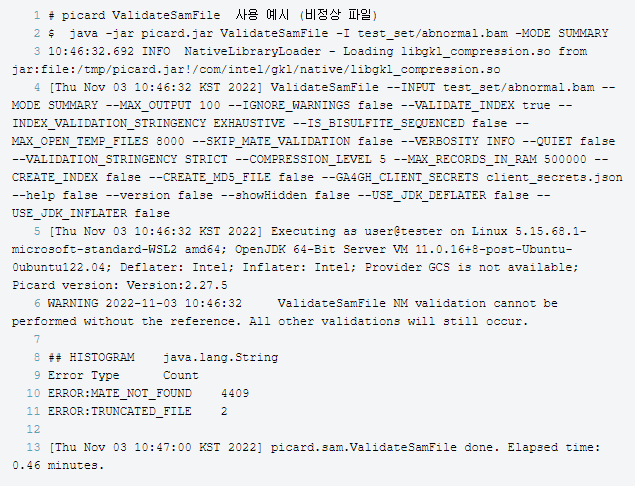
ValidateSamFile을 통해 출력되는 BAM 파일 포맷과 관련된 오류 항목과 이에 대한 설명은 여기서 확인하실 수 있습니다.
VCF 파일
VCF 파일은 변이 정보를 포함하고 있습니다. BCF라는 바이너리 파일로 변환하여 분석에 사용하기도 합니다. VCF 파일 포맷은 아래와 같습니다.

파일의 헤더 정보와 각 항목의 정보는 여기에 상세히 설명되어 있습니다. VCF 파일 포맷 검증 방법으로 GATK라는 분석 툴의 ValidateVariants라는 모듈이 있습니다. VCF 파일 포맷과 GVCF 파일 포맷 모두 검증을 할 수 있습니다. GATK는 2022년 11월 기준 4.3.0.0 버전을 제공하고 있습니다. JAR 파일로 구성되어 있어 JAVA 애플리케이션을 구동할 수 있도록 JRE 등을 설치 후 커맨드 라인으로 명령어를 입력하여 사용할 수 있습니다. GATK는 여기서 다운로드 할 수 있고 별도의 설치 과정 없이 다운로드 한 압축 파일을 다운로드 받아 압축을 해제하면 됩니다. 분석 옵션 목록과 상세 설명은 여기에서 확인하실 수 있습니다.
ValidateVariants의 사용 방법은 아래와 같습니다. 검증할 VCF 파일 경로와 VCF 생성에 사용된 참조서열 파일 경로를 입력해 줍니다. 참조서열 파일은 필수 입력 옵션은 아닙니다. VCF 파일만 입력하는 경우 파일 항목이 올바르게 입력되었는지, 중복되는 변이 정보는 없는지 등 파일 형식에 대한 검증만 수행하지만, 참조서열도 함께 입력하면 변이 정보가 유효한 참조서열 영역에 위치하는지 또는 위치별 REF 항목의 allele 정보가 참조서열과 같은 서열인지 등을 검증해 줍니다. --verbosity 옵션 프로그램 실행 중 또는 완료 후 출력되는 로그를 선택할 수 있는 옵션입니다.
# GATK ValidateVariants 사용 방법 $ gatk ValidateVariants -V <VCF File Path> -R <Reference Sequence Path> --verbosity <ERROR, WARNING,INFO, DEBUG>
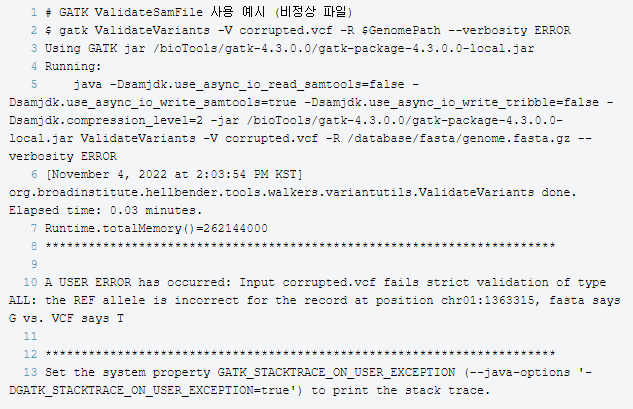
아래는 정상적인 VCF 파일 입력 파일로 지정한 경우입니다. 출력되는 로그는 아래와 같습니다.

아래는 비정상적인 VCF 파일을 이용한 경우입니다. 예제 파일은 REF allele 정보를 임의로 변경한 VCF 파일을 이용했습니다. 하단의 로그의 열 번째 줄을 보시면 A USER ERROR has occurred 로 시작하는 오류 로그를 확인할 수 있습니다.
마치며
NGS 분석에 활용되고 생성되는 대표적인 파일 포맷을 검증할 수 있는 툴을 소개해 드렸습니다. 물론 설명해 드린 세 개의 파일 포맷 이외에 언급하지 못한 다양한 파일 포맷과 다양한 툴들이 있습니다. 또한 python이나 R 등에서 관련 파일들을 편리하게 다룰 수 있는 library 등도 제공하고 있습니다. 소소하나마 시간을 절약할 수 있는 팁이 되었으면 좋겠습니다.
참고자료

EDITOR
박서우
Bioinformatics System Dept. · Senior Developer
'STORAGE' 카테고리의 다른 글
| 인실리코젠 프로젝트 : 남북 이산가족 찾기 (0) | 2022.12.26 |
|---|---|
| 비정형 유기체적 형상의 디자인 분야 활용 (0) | 2022.12.12 |
| 공공 SW 사업 제안요청서 변천 과정 (0) | 2022.11.15 |
| 식물과 미생물의 생존 본능, 지그재그 모델 (0) | 2022.10.31 |
| 음악의 뇌과학 (0) | 2022.10.11 |






댓글